青岛能源所发明简易高效的单细胞分选与测序对接技术
为了满足考察自然界中细胞“原位功能”这一共性科学需求,“现场”、“实时”的单细胞分选与测序已成为生命科学装备研制领域的一个重要发展趋势。尽管第三代测序技术已经实现了仪器微型化,但是与测序对接的单细胞精准分选装备却仍然相当笨重和昂贵,难以支撑各种科学考察中针对微生物组功能的“现场分析”。最近,青岛能源所单细胞研究中心马波研究员带领的微流控系统团队,通过设计简易高效的单细胞分选与测序对接装置,实现了每个试管有且只有一个细胞(“One-Cell-One-Tube”),有望服务于“现场”、“实时”乃至“便携式”的单细胞分选与测序。
细胞是地球上生命的功能单元和进化单位,因此单细胞分选与测序技术能够在最“深”的精度解析生命的功能机制与进化历程。在微生物世界中,各种生境下微生物细胞的“原位活性”深刻地塑造着地球生态系统,但这些活性在样品转移与运输过程中往往发生难以预测的变化,在实验室环境下也通常难以准确保存与严格复制,因此“现场”、“实时”的单细胞分选与测序已成为微生物组方法学领域的共性挑战。
与人体和高等动植物细胞相比,微生物细胞通常更小(0.1-10 微米),更加难于精准操纵,因此分离获取目标单细胞、并且实现测序反应要求的“One-Cell-One-Tube”是一个关键难点。目前的自动单细胞分离方法大多依赖于昂贵且体积庞大的荧光流式细胞分选仪(FACS);而现有的手动单细胞分离和测序方案,在依赖于操作人员熟练程度的同时,同样需要显微单细胞移液平台、激光光镊等同样难以随身携带的大中型仪器。此外,单细胞分选及核酸制备过程极易受到环境中飘浮微生物及其DNA的污染,而且这种污染通常难以在测序数据处理环节完全去除。因此,尽管目前MinION等第三代测序仪已经实现了便携化,微生物单细胞分选和测序仍然操作繁琐、污染干扰严重、而且难以满足“现场”、“实时”乃至“便携式”的要求。
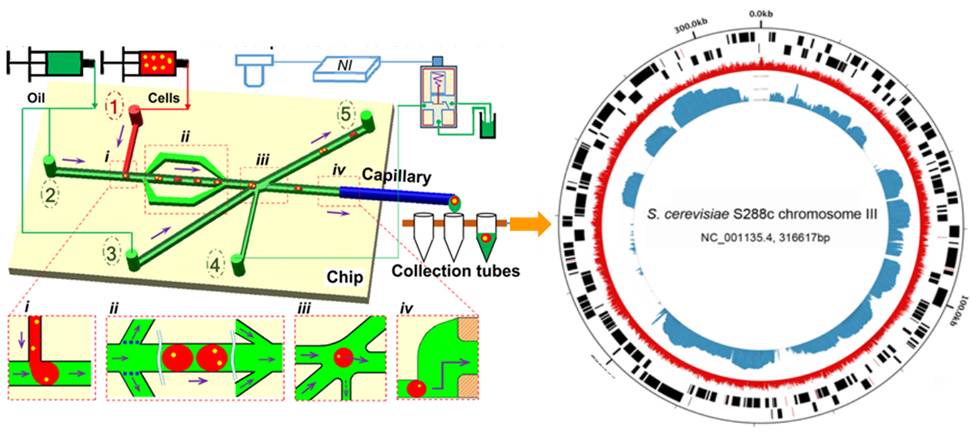
针对上述挑战,单细胞中心张强和王婷婷等发明了一种名为“FOCOT(Facile One-Cell-One-Tube)”的方法,能够精确、高速、低成本地分离、获取与分装单个微生物细胞,从而与单细胞测序直接对接。首先,通过微流控技术,将细胞分散包裹在数十微米直径的油包水微液滴中。其次,基于液滴显微光学成像识别技术,分选出单细胞包裹液滴。第三,将单细胞包裹液滴顺序分布于系列试管中,从而快速实现单个细胞的分离,以及每个试管有且只有一个细胞,以实现与单细胞全基因组扩增与测序的直接对接。
FOCOT平台主要有三个特色。第一,在简易方便方面,FOCOT平台除自行设计的芯片之外,仅需要电磁阀、平板电脑和普通光学显微镜,不需外接任何高成本商品化仪器平台,具有易获取、易替换、低成本等优势。同时,模块化、小型化、操作简便的设计使得该装置适合在自然环境实地采样条件下的携带、装配和使用,也几乎不需要额外的人员培训和技术维护,因此尤其适用于面向各种极端自然环境的科学考察,也有利于在普通实验室的推广应用。
第二,在分选高效方面,FOCOT平台通过显微镜下对包裹有单个细胞的液滴的准确识别和分选,能有效避免假阳性;而且其20秒每个的分选速度,与显微单细胞移液、激光光镊等现有的商品化分选装备相比具有明显优势。同时,单细胞获取率高于90%,培养成功比例至少80%,证明该方法能有效避免芯片表面吸附所导致的输运过程中细胞流失,而且对细胞活性没有或较小损伤。
第三,在污染控制方面,FOCOT平台涉及部件少,体积小型化,相对封闭,故在实验过程中,能够方便地实现超洁净环境空间控制、芯片消毒等操作,严格控制环境DNA的污染。对分离获取的单个酵母细胞进行全基因组扩增与测序后结果显示,99%的有效序列可以与参考基因组匹配,表明该方法能有效避免环境中微生物带来的DNA污染,平均基因组覆盖率达到43.3%,与在昂贵的超净空间设施中采用FACS等大型仪器系统分离获取单细胞所获得的测序结果相当。
目前,通过耦合FOCOT与中心前期开发的单细胞拉曼成像、拉曼流式细胞分选等技术,单细胞中心正在构建一个服务于岸基、船基乃至手基等不同需求的非标记式单细胞分析装备体系,以服务于能源、环境、健康、海洋、土壤等诸多微生物组应用领域。
上述工作由单细胞中心马波和徐健共同主持完成,获得了国家基金委科学仪器基础研究专项、面上项目和中科院生物高通量分析技术服务网络(STS)等项目的支持。
附录:
Qiang Zhang, Tingting Wang, Qian Zhou, Peng Zhang, Yanhai Gong, Honglei Gou, Jian Xu, and Bo Ma, Development of a facile droplet-based single-cell isolation platform for cultivation and genomic analysis in microorganisms. Sci Rep, 7:41192, DOI: 10.1038/srep41192
附件下载: