青岛能源所在计算机辅助蛋白质设计取得新进展
蛋白质作为药物、生物制剂和催化剂被广泛应用,其稳定性对于生物技术应用具有重要影响。通常,蛋白质的稳定性可以通过定向进化、序列同源比对和理性设计来完成。上述方法都有各自优势和缺点,在实际应用中方法的适用性和成功率通常依赖于蛋白质体系本身。
近日,中国科学院青岛生物能源与过程研究所仿真模拟团队设计出一套计算机虚拟筛选结合分子生物学实验方法,成功提高了枯草芽孢杆菌蓝色荧光蛋白质YtVA LOV的稳定性。该方法主要包括如下步骤:1.采用计算机模拟对可能的热稳定突变进行筛选;2.对筛选出较重要突变体进行实验验证;3.用计算模拟对实验确认的位点及其周围进行氨基酸再优化;4.对优化的突变位点进行实验表征;5.对获取的稳定突变进行组合。实验中,采用该方法成功将YtVA LOV的Tm值提高了31°C(图1)。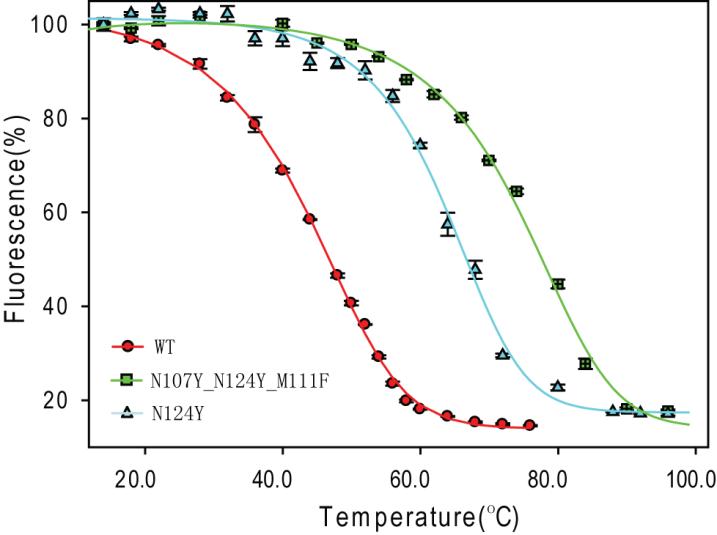
图1.YtVA LOV荧光强度随温度的变化曲线。
该方法的优势在于不受蛋白质体系本身限制,理论上可以用来提高任何具有三维结构的蛋白质的稳定性,对稳定性差的蛋白质制剂改性具有重要意义。该研究成果已经在线发表于Plos Computational Biology。
原文链接:(免费下载)
http://www.ploscompbiol.org/article/info:doi/10.1371/journal.pcbi.1003129
(仿真模拟团队供稿)
附件下载: