青岛能源所开发物种-代谢双靶向的微生物细胞及酶资源挖掘新技术
微生物及其合成的各种酶支撑着生物圈中诸多关键的生态过程。如何从环境中高效地识别与挖掘具有特定原位代谢功能的细胞和酶,是微生物组科学与产业长期以来的一个热点和难点。近日,青岛能源所联合自然资源部第一海洋研究所、山东大学、青岛星赛生物科技有限公司等单位,合作开发了FISH-scRACS-seq(荧光原位杂交介导的拉曼激活单细胞分选与测序)技术,能够“物种-代谢”双靶向性地从环境样品中直接识别和挖掘功能单细胞及其编码的酶资源。研究人员利用该技术成功识别和分选出海洋中活跃降解环烷烃的γ-变形菌,进而通过其单细胞全基因组序列,发现了一类在全球低温海洋中降解环烷烃的P450酶。FISH-scRACS-seq能从全生态系统的范围,以单个菌体的精度,建立生态过程特征、细胞原位代谢能力、全基因组序列、代谢途径、酶催化功能这五个生命尺度之间的关联机制,从而为微生物及其酶资源的发现和挖掘开辟了一条新颖的技术路线。相关研究成果发表于《创新(英文)》 (The Innovation)。
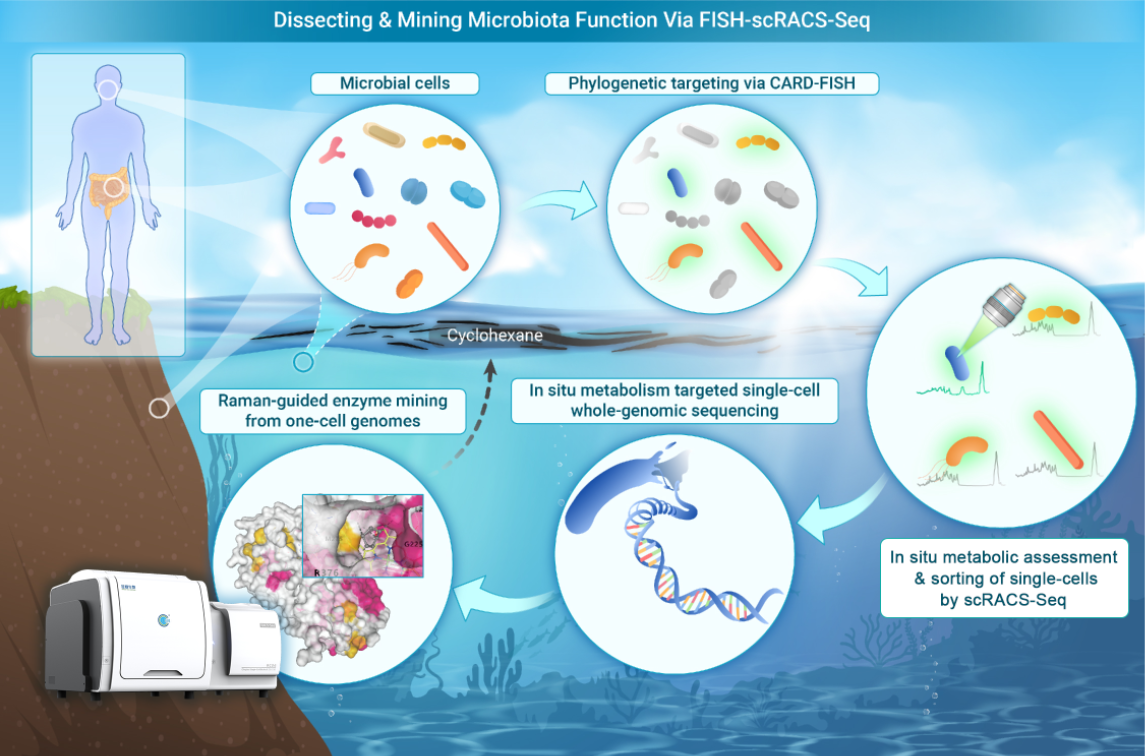
利用FISH-scRACS-Seq剖析并挖掘微生物组功能
当前,微生物功能挖掘主要依赖两种策略。一种是基因型驱动策略,如基于元基因组测序和比较的微生物组关联分析(MWAS)。它能够揭示诸多与生态过程特征相关联的候选微生物类群,但通常难以揭示这些微生物类群的原位代谢功能及其背后的分子机制。这主要是因为验证这些目标微生物类群的原位代谢活动、并将这些活动追溯到完整的基因组尚存在很多技术瓶颈。另一种是代谢表型驱动策略,如拉曼激活细胞分选与单细胞测序技术(RACS-seq)。其可通过拉曼光谱直接测量和分选微生物组中具有目标原位代谢功能的单细胞,并获取其全基因组或纯培养物。然而,一个环境样本通常含有海量的微生物细胞,而其中的目标代谢功能菌往往相对丰度很低。因此,利用RACS-seq技术直接从环境中识别目标代谢功能菌受限于其通量和成本。同时,并非所有具原位代谢功能的微生物均对关注的生态过程特征作出贡献。因此,如何针对MWAS发现的对生态过程特征有贡献的目标微生物物种,从环境样品中直接识别和挖掘其全基因组及其编码的酶资源,是业界面临的共性方法学挑战。
针对上述瓶颈问题,青岛能源所单细胞中心的高级工程师荆晓艳、副研究员公衍海、刁志钿、马燕等组成联合研究小组,开发了一种“双管齐下”的FISH-scRACS-seq技术。该方法先利用荧光原位杂交(FISH),基于荧光标记的物种靶向性DNA序列杂交探针,识别出菌群中目标物种的微生物细胞。在此基础上,通过单细胞拉曼光谱表征这些目标物种细胞的原位代谢功能,进而“物种-代谢”双靶向、一一对应地分选出目标物种和目标代谢功能的单细胞并测定其全基因组序列,从而解析其代谢机制并获取基因组上编码的功能元件。
通过纯培养混菌体系和复杂土壤菌群,研究小组首先验证了FISH-scRACS-seq技术的特异性和灵敏度。经荧光原位杂交和单细胞拉曼信号采集、双靶向分选出的目标功能单细胞,其基因组覆盖度最高可达99.14%。这样的高覆盖度对于在该基因组上穷尽式地挖掘能解释和支撑该细胞原位代谢功能的酶基因或调控元件至关重要。
环烷烃在地下碳氢化合物储层和气体凝析油中丰富,对水生生物具有高度毒性,并且难以降解,因此在大规模石油泄漏发生时,环烷烃会带来显著的生态风险。尽管对海洋生态系统中环烷烃的生物降解机制知之甚少,但最近的研究已通过MWAS测序,将一组未培养的、嗜冷和寡营养的γ-变形菌与中国边缘海的环烷烃降解特性相关联。因此,从渤海天然气凝析油田附近的海水出发,研究小组针对这类γ-变形菌的基因组设计了FISH探针。进而,通过环己烷饲喂耦合重水代谢示踪、荧光成像、拉曼成像和单细胞拉曼分选,测定并分选出了那些展现原位降解环己烷代谢能力的γ-变形菌细胞,并解析了其单细胞基因组。他们发现了一种原位高效降解环己烷的假交替单胞菌(Pseudoalteromonas fuliginea),这是首次发现假交替单胞菌具有降解环己烷的“超能力”。
基因组分析表明,这种假交替单胞菌利用一种新型的细胞色素P450酶系统(P450PsFu)来降解环己烷分子。体外酶活实验和气相色谱-质谱(GC-MS)分析表明,该酶系统能够在体外将环己烷(有生物毒性)转化为环己醇(无生物毒性),这是环烷烃降解的第一步,也是限速步骤。P450PsFu属于CYP236A亚家族的P450酶,该类P450酶之前仅被认为可特异性降解6-O-甲基-D-半乳糖,因此这是一类前所未知的、具有降解环己烷催化活性的P450酶。这类酶在全球海洋微生物组中虽然很少见、相对含量也很低,但却具全球分布特征,且大多出现在极地等低温海域。这些原位降解环烷烃的微生物与酶的发现,拓展了低温海洋生态系统中环烷烃降解的认知视角,也为烃类污染海洋环境的生物修复提供了新资源。这些发现也表明,FISH-scRACS-seq与上游MWAS相结合,为建立从生态过程特征到细胞与酶的催化功能之间的关联机制,提供了一个具有共性应用价值的解决方案。
该研究由青岛能源所单细胞中心徐健研究员、山东大学马莉副教授、自然资源部第一海洋研究所崔志松研究员指导完成,得到了国家重点研发计划青年科学家项目和国家自然科学基金项目等的资助。
原文链接:https://www.sciencedirect.com/science/article/pii/S2666675824001978
Xiaoyan Jing#,Yanhai Gong#,Zhidian Diao#,Yan Ma#,Yu Meng,Jie Chen,Yishang Ren,Yuting Liang,Yinchao Li,Weihan Sun,Jia Zhang,Yuetong Ji,Zhiqi Cong,Shengying Li,Bo Ma,Zhisong Cui*,Li Ma*,Jian Xu*. Phylogeny-metabolism dual-directed single-cell genomics for dissecting and mining ecosystem function by FISH-scRACS-seq. The Innovation, 2025.
附件下载: